жқҘжәҗ I еҲҶеӯҗи®ҫи®ЎпјҲID:gh_4a8e2c6cbed4пјү
2023е№ҙ4жңҲ14ж—ҘпјҢе®ҮйҒ“з”ҹзү©пјҲNutshell BiotechпјүиҒ”еҗҲдёҠжө·дәӨйҖҡеӨ§еӯҰеӣўйҳҹеңЁзҫҺеӣҪеҢ–еӯҰдјҡжңҹеҲҠJournal of Chemical Information and ModelingдёҠеҸ‘иЎЁдәҶйўҳдёәвҖңResearch and Evaluation of the Allosteric Protein-Specific Force Field Based on a Pre-Training Deep Learning ModelвҖқзҡ„и®әж–ҮгҖӮиҜҘз ”з©¶жҸҗеҮәеҹәдәҺиӣӢзҷҪеҸҳжһ„ж•Ҳеә”ејҖеҸ‘дё“жңүеҠӣеңәзҡ„жҰӮеҝөпјҢ并иҺ·еҫ—дәҶйҰ–дёӘйҖӮз”ЁдәҺжҚ•жҚүиӣӢзҷҪеҸҳжһ„ж•Ҳеә”зҡ„еҲҶеӯҗеҠӣеңәпјҢе‘ҪеҗҚдёәAPSFпјҲAllostericВ ProteinВ SpecificВ Force FieldпјүгҖӮ
 еңЁз”ҹзү©дҪ“еҶ…пјҢиӣӢзҷҪзӯүз”ҹзү©еӨ§еҲҶеӯҗе§Ӣз»Ҳж—¶еҲ»еҠЁжҖҒеҸҳеҢ–пјҢ并йҡҸе…¶еҠҹиғҪжүҖйңҖиҝӣиЎҢжһ„иұЎи°ғж•ҙпјҢж¶үеҸҠеҸҳеҢ–е°әеәҰд»Һи§ЈжҠҳеҸ -жҠҳеҸ еӨ§и§„жЁЎеӨ–йғЁиҝҗеҠЁпјҢеҲ°еҲҶеӯҗе№Ійў„дёӯзҡ„еҶ…йғЁзІҫз»ҶиҜұеҜјеҘ‘еҗҲгҖӮеҸҳжһ„пјҲAllosteric regulationпјүжҳҜз”ҹзү©еӨ§еҲҶеӯҗжҷ®йҒҚеӯҳеңЁзҡ„дёҖз§Қз”ҹзү©еӯҰи°ғжҺ§ж–№ејҸпјҢйҖҡиҝҮеә•зү©жҙ»жҖ§дҪҚзӮ№еӨ–зҡ„еҸҳжһ„дҪҚзӮ№пјҲAllosteric siteпјүе№Ійў„жқҘи§ҰеҸ‘жһ„иұЎеҠЁжҖҒеҸҳеҢ–пјҢе®һзҺ°еҗ‘дёҠжҲ–еҗ‘дёӢеҠҹиғҪж”№еҸҳгҖӮ2010е№ҙд»ҘжқҘпјҢйҡҸзқҖASDеӨ§ж•°жҚ®й©ұеҠЁзҡ„еҸҳжһ„жңәеҲ¶з ”究е’Ңж–№жі•зӘҒз ҙпјҢеҸҳжһ„дҪҚзӮ№и°ғжҺ§жңүжңӣжҲҗдёәдёҖз§ҚиҚҜзү©и®ҫи®ЎжҠҖжңҜпјҢйқўеҗ‘й…¶жҝҖеҠЁеүӮгҖҒ家ж—ҸиӣӢзҷҪдәҡеһӢйҖүжӢ©жҖ§гҖҒиӣӢзҷҪдә’дҪңеҸҠиҖҗиҚҜеҸ‘з”ҹзҡ„йҡҫжҲҗиҚҜйқ¶ж ҮпјҢиҺ·еҫ—дәҶеӨҡдёӘдёҠеёӮеҸҳжһ„иҚҜзү©зҡ„йӘҢиҜҒпјҢеҰӮAsciminibзӯүгҖӮ然иҖҢпјҢе·ІеҸ‘зҺ°жҙ»жҖ§еҲҶеӯҗеӨ§еӨҡжқҘиҮӘйҡҸжңәеҸ‘зҺ°пјҢеҸҜз”ЁдәҺеҲҶеӯҗи®ҫи®Ўзҡ„еҸҳжһ„дҪҚзӮ№е°‘жңүиҜҶеҲ«пјҢе°Өе…¶жҳҜеңЁиӣӢзҷҪжһ„иұЎеҠЁжҖҒеҸҳеҢ–дёӯжҲҗиҚҜжҖ§иҫғеҘҪзҡ„йҡҗејҸеҸҳжһ„дҪҚзӮ№пјҲHidden/Cryptic allosteric siteпјүпјҢжӣҙжҳҜиҪ¬зһ¬еҚійҖқйҡҫд»ҘжҚ•жҚүгҖӮеңЁиӣӢзҷҪзӯүз”ҹзү©еӨ§еҲҶеӯҗзҡ„еҠЁжҖҒеҸҳеҢ–з ”з©¶дёӯпјҢеҲҶеӯҗеҠЁеҠӣеӯҰпјҲMolecular dynamics simulationsпјҢMDпјүжҳҜдёҖз§ҚжЁЎжӢҹз”ҹзү©еӨ§еҲҶеӯҗеңЁжә¶ж¶ІдёӯеҸҳеҢ–规еҫӢзҡ„и®Ўз®—ж–№жі•пјҢйҖҡиҝҮеҲҶеӯҗеҠӣеңәпјҲForce fieldпјүй©ұеҠЁз”ҹзү©еҲҶеӯҗиҝҗеҠЁпјҢд»ҺиҖҢеҸ‘зҺ°е®һйӘҢжүҖдёҚиғҪиҺ·еҫ—зҡ„дёҖдәӣжһ„иұЎгҖӮз”ұдәҺиӣӢзҷҪеңЁеҸҳжһ„иҝҮзЁӢдёӯдёҚд»…еҸ‘з”ҹеҲҶеӯҗеҶ…йҖҡи®ҜпјҢиҖҢдё”жһ„иұЎеҸҳеҢ–дә§з”ҹзҡ„дҪҚзӮ№з©әи…”еҮәзҺ°жӣҙдёәзҹӯжҡӮпјҢйҖ жҲҗдј з»ҹеҠӣеңәйҡҫд»ҘжҚ•жҚүпјҢеҰӮиғҪй’ҲеҜ№еҸҳжһ„ж•Ҳеә”зҡ„дёҠиҝ°зү№еҫҒеҺ»ејҖеҸ‘дё“жңүеҲҶеӯҗеҠӣеңәпјҢдёҚд»…жңүеҠ©дәҺйҳҗжҳҺеҸҳжһ„жңәеҲ¶е№¶жҚ•жҚүйҡҗејҸеҸҳжһ„дҪҚзӮ№пјҢжӣҙдёәйҮҚиҰҒзҡ„жҳҜпјҢдёәйҡҫйқ¶йқ¶ж Үзҡ„еҺҹеҲӣж–°иҚҜзӘҒз ҙжҸҗдҫӣдәҶиҚҜзү©и®ҫи®Ўзҡ„з ”еҸ‘жәҗеӨҙгҖӮ
еңЁз”ҹзү©дҪ“еҶ…пјҢиӣӢзҷҪзӯүз”ҹзү©еӨ§еҲҶеӯҗе§Ӣз»Ҳж—¶еҲ»еҠЁжҖҒеҸҳеҢ–пјҢ并йҡҸе…¶еҠҹиғҪжүҖйңҖиҝӣиЎҢжһ„иұЎи°ғж•ҙпјҢж¶үеҸҠеҸҳеҢ–е°әеәҰд»Һи§ЈжҠҳеҸ -жҠҳеҸ еӨ§и§„жЁЎеӨ–йғЁиҝҗеҠЁпјҢеҲ°еҲҶеӯҗе№Ійў„дёӯзҡ„еҶ…йғЁзІҫз»ҶиҜұеҜјеҘ‘еҗҲгҖӮеҸҳжһ„пјҲAllosteric regulationпјүжҳҜз”ҹзү©еӨ§еҲҶеӯҗжҷ®йҒҚеӯҳеңЁзҡ„дёҖз§Қз”ҹзү©еӯҰи°ғжҺ§ж–№ејҸпјҢйҖҡиҝҮеә•зү©жҙ»жҖ§дҪҚзӮ№еӨ–зҡ„еҸҳжһ„дҪҚзӮ№пјҲAllosteric siteпјүе№Ійў„жқҘи§ҰеҸ‘жһ„иұЎеҠЁжҖҒеҸҳеҢ–пјҢе®һзҺ°еҗ‘дёҠжҲ–еҗ‘дёӢеҠҹиғҪж”№еҸҳгҖӮ2010е№ҙд»ҘжқҘпјҢйҡҸзқҖASDеӨ§ж•°жҚ®й©ұеҠЁзҡ„еҸҳжһ„жңәеҲ¶з ”究е’Ңж–№жі•зӘҒз ҙпјҢеҸҳжһ„дҪҚзӮ№и°ғжҺ§жңүжңӣжҲҗдёәдёҖз§ҚиҚҜзү©и®ҫи®ЎжҠҖжңҜпјҢйқўеҗ‘й…¶жҝҖеҠЁеүӮгҖҒ家ж—ҸиӣӢзҷҪдәҡеһӢйҖүжӢ©жҖ§гҖҒиӣӢзҷҪдә’дҪңеҸҠиҖҗиҚҜеҸ‘з”ҹзҡ„йҡҫжҲҗиҚҜйқ¶ж ҮпјҢиҺ·еҫ—дәҶеӨҡдёӘдёҠеёӮеҸҳжһ„иҚҜзү©зҡ„йӘҢиҜҒпјҢеҰӮAsciminibзӯүгҖӮ然иҖҢпјҢе·ІеҸ‘зҺ°жҙ»жҖ§еҲҶеӯҗеӨ§еӨҡжқҘиҮӘйҡҸжңәеҸ‘зҺ°пјҢеҸҜз”ЁдәҺеҲҶеӯҗи®ҫи®Ўзҡ„еҸҳжһ„дҪҚзӮ№е°‘жңүиҜҶеҲ«пјҢе°Өе…¶жҳҜеңЁиӣӢзҷҪжһ„иұЎеҠЁжҖҒеҸҳеҢ–дёӯжҲҗиҚҜжҖ§иҫғеҘҪзҡ„йҡҗејҸеҸҳжһ„дҪҚзӮ№пјҲHidden/Cryptic allosteric siteпјүпјҢжӣҙжҳҜиҪ¬зһ¬еҚійҖқйҡҫд»ҘжҚ•жҚүгҖӮеңЁиӣӢзҷҪзӯүз”ҹзү©еӨ§еҲҶеӯҗзҡ„еҠЁжҖҒеҸҳеҢ–з ”з©¶дёӯпјҢеҲҶеӯҗеҠЁеҠӣеӯҰпјҲMolecular dynamics simulationsпјҢMDпјүжҳҜдёҖз§ҚжЁЎжӢҹз”ҹзү©еӨ§еҲҶеӯҗеңЁжә¶ж¶ІдёӯеҸҳеҢ–规еҫӢзҡ„и®Ўз®—ж–№жі•пјҢйҖҡиҝҮеҲҶеӯҗеҠӣеңәпјҲForce fieldпјүй©ұеҠЁз”ҹзү©еҲҶеӯҗиҝҗеҠЁпјҢд»ҺиҖҢеҸ‘зҺ°е®һйӘҢжүҖдёҚиғҪиҺ·еҫ—зҡ„дёҖдәӣжһ„иұЎгҖӮз”ұдәҺиӣӢзҷҪеңЁеҸҳжһ„иҝҮзЁӢдёӯдёҚд»…еҸ‘з”ҹеҲҶеӯҗеҶ…йҖҡи®ҜпјҢиҖҢдё”жһ„иұЎеҸҳеҢ–дә§з”ҹзҡ„дҪҚзӮ№з©әи…”еҮәзҺ°жӣҙдёәзҹӯжҡӮпјҢйҖ жҲҗдј з»ҹеҠӣеңәйҡҫд»ҘжҚ•жҚүпјҢеҰӮиғҪй’ҲеҜ№еҸҳжһ„ж•Ҳеә”зҡ„дёҠиҝ°зү№еҫҒеҺ»ејҖеҸ‘дё“жңүеҲҶеӯҗеҠӣеңәпјҢдёҚд»…жңүеҠ©дәҺйҳҗжҳҺеҸҳжһ„жңәеҲ¶е№¶жҚ•жҚүйҡҗејҸеҸҳжһ„дҪҚзӮ№пјҢжӣҙдёәйҮҚиҰҒзҡ„жҳҜпјҢдёәйҡҫйқ¶йқ¶ж Үзҡ„еҺҹеҲӣж–°иҚҜзӘҒз ҙжҸҗдҫӣдәҶиҚҜзү©и®ҫи®Ўзҡ„з ”еҸ‘жәҗеӨҙгҖӮ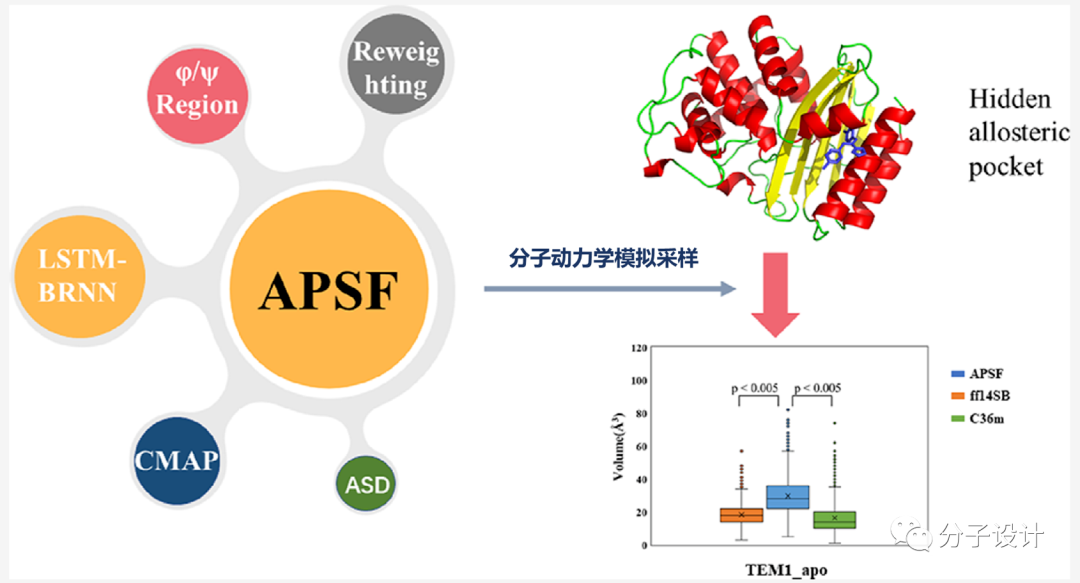 й’ҲеҜ№иҝҷдёҖйҮҚеӨ§з§‘еӯҰй—®йўҳпјҢе®ҮйҒ“з”ҹзү©дёҺдёҠжө·дәӨйҖҡеӨ§еӯҰзҡ„科еӯҰ家еӣўйҳҹд»ҺеҲҶеӯҗеҠӣеңәеә•еұӮзҹҘиҜҶжЎҶжһ¶еҮәеҸ‘пјҢеҹәдәҺе®ҮйҒ“AlloMatrixеҸҳжһ„дҪ“зі»ж•°жҚ®пјҢйҖҡиҝҮDeep Learningдәәе·ҘжҷәиғҪз®—жі•иҝӣиЎҢеӨ§и§„жЁЎеҠӣеңәеҸӮж•°дјҳеҢ–пјҢеҸ‘еұ•еҲӣе»әдәҶеҸҳжһ„зү№ејӮжҖ§еҠӣеңәAPSFпјҢеңЁPDK1гҖҒCDK2гҖҒEGFRгҖҒEEDзӯү8дёӘдҪ“зі»жөӢиҜ•дёӯпјҢжңүж•ҲжҚ•жҚүеҲ°еҸҳжһ„ж•Ҳеә”пјҢжҲҗеҠҹдәҶеӨҚзҺ°иҝҷдәӣдҪ“зі»дёӯзҡ„жө…еұӮеҸҳжһ„дҪҚзӮ№пјҢ并жҢ–жҺҳеҮәдј з»ҹеҠӣеңәйҡҫд»ҘиҜҶеҲ«зҡ„йҡҗи—ҸеҸҳжһ„дҪҚзӮ№е’Ңж·ұеұӮеҸҳжһ„дҪҚзӮ№пјҢе®һзҺ°дәҶеҜ№еҸҳжһ„ж•Ҳеә”зҡ„й«ҳж•ҲзІҫеҮҶеҸ‘зҺ°гҖӮ
еңЁеҜ№еҠӣеңәзҡ„зі»з»ҹжөӢиҜ•дёӯпјҢзӣёиҫғдәҺдј з»ҹAmberе’ҢCharmmеҠӣеңәпјҢAPSFеҜ№иӣӢзҷҪдҪ“зі»зҡ„еҸҳжһ„жһ„иұЎйҮҮж ·ж•ҲзҺҮжӣҙй«ҳгҖҒеҜ№еҸҳжһ„дҪҚзӮ№еҮәзҺ°жҚ•жҚүзҡ„зЁӢеәҰжӣҙеҮҶпјҢиЎЁжҳҺиҜҘеҠӣеңәдёҚд»…жңүж•ҲжҸҗеҚҮиӣӢзҷҪеҸҳжһ„жңәеҲ¶зҡ„жҺўзҙўе’Ңз ”з©¶пјҢд№ҹеҸҜз”ЁдәҺдҪҚзӮ№иҜҶеҲ«еҸҠеҸҳжһ„еҲҶеӯҗеҸ‘зҺ°пјҢдёәеҸҳжһ„жһ„иұЎй©ұеҠЁзҡ„иҚҜзү©и®ҫи®Ўж–№жі•еҸ‘еұ•еҘ е®ҡдәҶеқҡе®һзҡ„еә•еұӮз”ҹзү©еӨ§еҲҶеӯҗзҗҶи®әи®Ўз®—еҹәзЎҖгҖӮ
Xiaoyue Ji, Xiaochen Cui, Zhengxin Li, Taeyoung Choi, Ying Wang, Wen Xiao, Yunshuo Zhao, Jinyin Zha, Jian Zhang, Hai-Feng Chen, Zhengtian Yu. Research and Evaluation of the Allosteric Protein-Specific Force Field Based on a Pre-Training Deep Learning Model. J Chem Inf Model. 2023, doi: 10.1021/acs.jcim.2c01369
й’ҲеҜ№иҝҷдёҖйҮҚеӨ§з§‘еӯҰй—®йўҳпјҢе®ҮйҒ“з”ҹзү©дёҺдёҠжө·дәӨйҖҡеӨ§еӯҰзҡ„科еӯҰ家еӣўйҳҹд»ҺеҲҶеӯҗеҠӣеңәеә•еұӮзҹҘиҜҶжЎҶжһ¶еҮәеҸ‘пјҢеҹәдәҺе®ҮйҒ“AlloMatrixеҸҳжһ„дҪ“зі»ж•°жҚ®пјҢйҖҡиҝҮDeep Learningдәәе·ҘжҷәиғҪз®—жі•иҝӣиЎҢеӨ§и§„жЁЎеҠӣеңәеҸӮж•°дјҳеҢ–пјҢеҸ‘еұ•еҲӣе»әдәҶеҸҳжһ„зү№ејӮжҖ§еҠӣеңәAPSFпјҢеңЁPDK1гҖҒCDK2гҖҒEGFRгҖҒEEDзӯү8дёӘдҪ“зі»жөӢиҜ•дёӯпјҢжңүж•ҲжҚ•жҚүеҲ°еҸҳжһ„ж•Ҳеә”пјҢжҲҗеҠҹдәҶеӨҚзҺ°иҝҷдәӣдҪ“зі»дёӯзҡ„жө…еұӮеҸҳжһ„дҪҚзӮ№пјҢ并жҢ–жҺҳеҮәдј з»ҹеҠӣеңәйҡҫд»ҘиҜҶеҲ«зҡ„йҡҗи—ҸеҸҳжһ„дҪҚзӮ№е’Ңж·ұеұӮеҸҳжһ„дҪҚзӮ№пјҢе®һзҺ°дәҶеҜ№еҸҳжһ„ж•Ҳеә”зҡ„й«ҳж•ҲзІҫеҮҶеҸ‘зҺ°гҖӮ
еңЁеҜ№еҠӣеңәзҡ„зі»з»ҹжөӢиҜ•дёӯпјҢзӣёиҫғдәҺдј з»ҹAmberе’ҢCharmmеҠӣеңәпјҢAPSFеҜ№иӣӢзҷҪдҪ“зі»зҡ„еҸҳжһ„жһ„иұЎйҮҮж ·ж•ҲзҺҮжӣҙй«ҳгҖҒеҜ№еҸҳжһ„дҪҚзӮ№еҮәзҺ°жҚ•жҚүзҡ„зЁӢеәҰжӣҙеҮҶпјҢиЎЁжҳҺиҜҘеҠӣеңәдёҚд»…жңүж•ҲжҸҗеҚҮиӣӢзҷҪеҸҳжһ„жңәеҲ¶зҡ„жҺўзҙўе’Ңз ”з©¶пјҢд№ҹеҸҜз”ЁдәҺдҪҚзӮ№иҜҶеҲ«еҸҠеҸҳжһ„еҲҶеӯҗеҸ‘зҺ°пјҢдёәеҸҳжһ„жһ„иұЎй©ұеҠЁзҡ„иҚҜзү©и®ҫи®Ўж–№жі•еҸ‘еұ•еҘ е®ҡдәҶеқҡе®һзҡ„еә•еұӮз”ҹзү©еӨ§еҲҶеӯҗзҗҶи®әи®Ўз®—еҹәзЎҖгҖӮ
Xiaoyue Ji, Xiaochen Cui, Zhengxin Li, Taeyoung Choi, Ying Wang, Wen Xiao, Yunshuo Zhao, Jinyin Zha, Jian Zhang, Hai-Feng Chen, Zhengtian Yu. Research and Evaluation of the Allosteric Protein-Specific Force Field Based on a Pre-Training Deep Learning Model. J Chem Inf Model. 2023, doi: 10.1021/acs.jcim.2c01369



